RからMplusを使う
構造方程式モデリングのためのソフトウェアと言えばMplusです。Mplusはシンタックスも簡単で計算も早くてとても良いソフトウェアですがアウトプットが見辛い上に,モデルの数だけインプットとアウトプットのファイルが増えていくのでごちゃつきます。また,私だけかもしれませんが,あとで解析をやりなおそうと思った際に,どのファイルを使ったのか分からなくなりがちです。
そこで,MplusをRから使えるパッケージとしてMplusAutomationというものがあります。日本語で解説している資料としては徳岡先生(高松大)によるものあり大変参考になります。
https://www.slideshare.net/masarutokuoka/rmplusmplusautomation-hiroshimar05
以下ではそのスライドを参考にしたり,パッケージのvignetteを見ながら試した記録を残します(主に将来の自分に向けて)。
モデルをRで書く
mplusObjectという関数を使うと,Mplusのinputファイルを用意できるようです。以下ではpsychパッケージ所収のbfiデータを使っていきます。とりあえず,5因子のCFAのモデルを書いてみます。
library(psych)
library(MplusAutomation)
test1 <- mplusObject(MODEL ="F1 BY A1-A5;
F2 BY C1-C5;
F3 BY E1-E5;
F4 BY N1-N5;
F5 BY O1-O5",
rdata = bfi)
res <- mplusModeler(test,
dataout = "test1.tsv",
modelout = "test1.inp", run = 1L)mplusObject関数の引数は,通常のMplusのsyntaxのセクションに書くような内容を書いていきます。上の例では,モデルの部分だけ書きましたが,書いていないVARIABLEのセクションなどは,MODELの部分から必要なものを選んで勝手に作ってくれているようです。
こうしてできたR上のMplus Model Objectを実行するためのものがmplusModeler関数です。第1の引数には先ほどのMplus Model Objectを指定します。実行すると,Model Objectを元にして,同じディレクトリ内に,Mplusのinput,Mplusの実行用のデータファイル,Mplusのアウトプットが生成されます。dataoutという引数には,Mplus実行用に生成されたデータのファイル名を指定します。このファイルでは,例えば,列名のヘッダーが削除されていたり,欠損値が「.」に置き換えられていたりと,Mplusで処理できる形にデータが変換されています。またmodeloutという引数には,モデルを実行するのに使われたMplusのインプットファイルのファイル名を渡します。runという引数に0を渡すとモデルは実行されず,Mplus用のデータファイルとインプットファイルだけが生成されるようです。モデルが意図した通りに.inpファイルに反映されているかどうかを確認したい時に使うもののようです。1より大きい場合にはブートストラップで繰り返すとのことです。
上の例では,とりあえず実行できれば良いということでモデルのみ書きましたが,もう少しいろいろと指定したものを作ってみます。
test2 <- mplusObject(
TITLE = "This is mplusautomation test 2;",
ANALYSIS = "ESTIMATOR = MLR;",
OUTPUT = "SAMPSTAT STDYX MODINDICES(ALL);",
VARIABLE = "USEVARIABLES = A1-A5 C1-C5 E1-E5 N1-N5 O1-O5;",
MODEL ="F1 BY A1-A5*;
F2 BY C1-C5*;
F3 BY E1-E5*;
F4 BY N1-N5*;
F5 BY O1-O5*;
F1@1 F2@1 F3@1 F4@1 F5@1;",
rdata = bfi)
res2 <- mplusModeler(test2,
dataout = "test2.tsv",
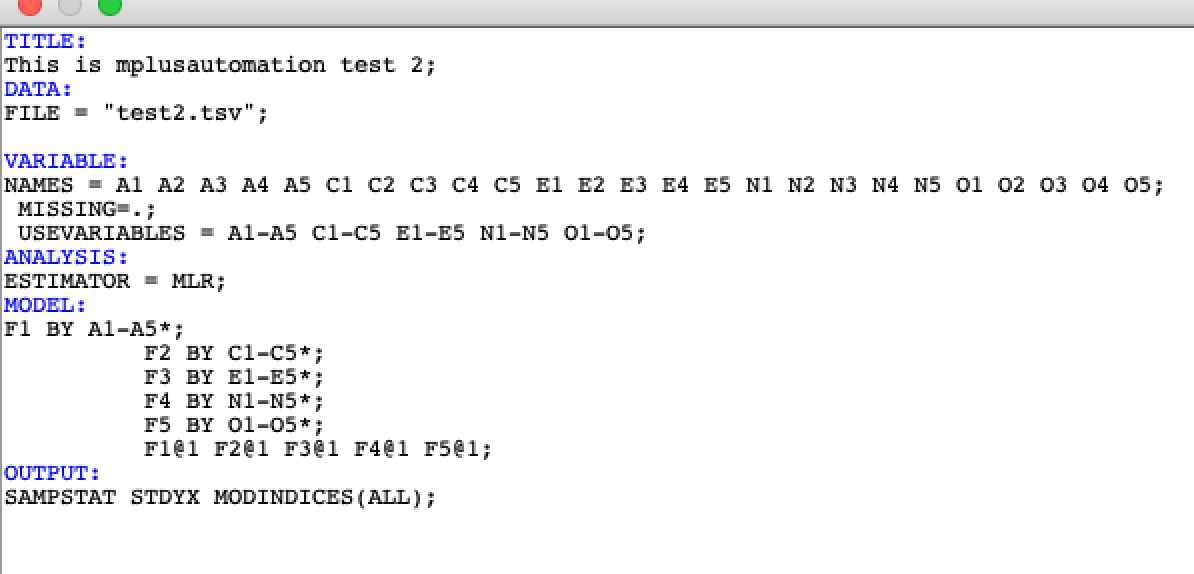
modelout = "test2.inp", run = 1L)以下の画像は,上の関数で作った.inpファイルです。mplusObjectで指定した情報が,.inpの各セクションに反映されていることがわかります。
結果をR上で見る
分析したファイルの結果をみたい場合には,結果のオブジェクトをsummary関数に渡せば良いようです。
summary(res2)
# readModels("test2.out", what = "summaries") アウトプットファイルを読み込ませても同じ出力が得られる
# res2$results$summariesでも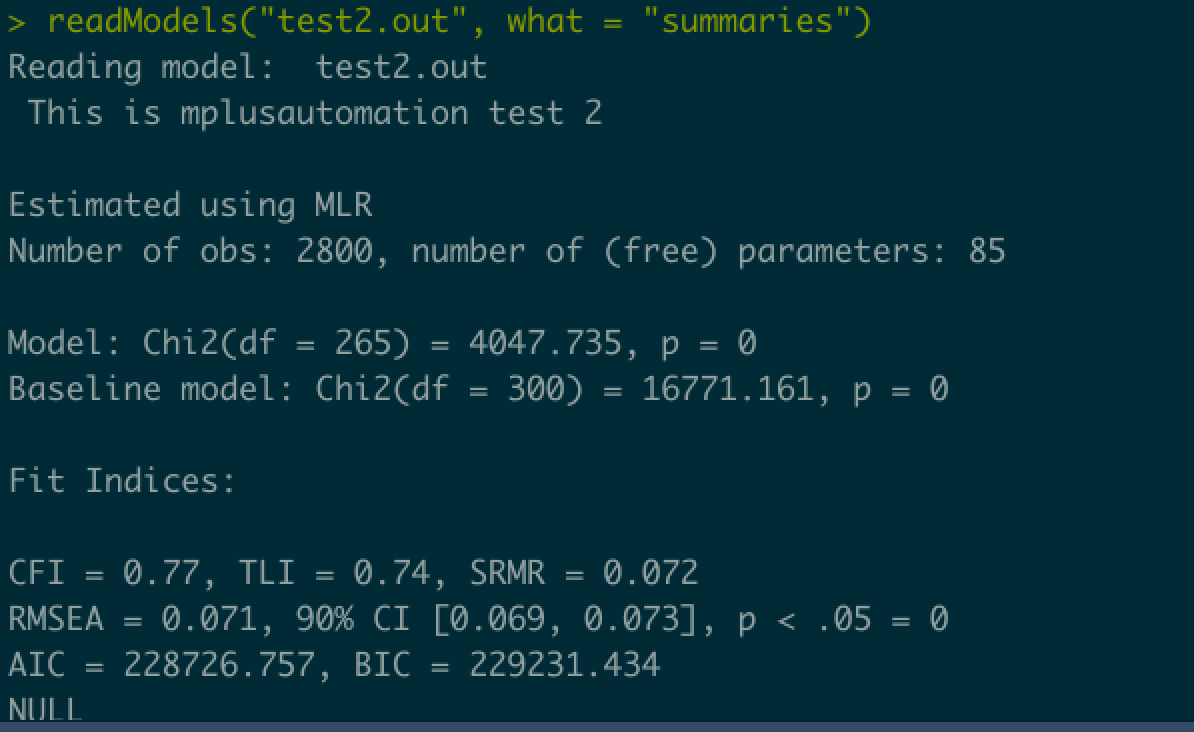
推定したパラメータをみたい場合には以下のように,Rのリストオペレータを使うか,readModels関数にアウトプットファイルを渡すと良いようです。
res2$results$parameters
# readModels("test2.out", what = "parameters")$parameters探索的因子分析の場合
同じデータセットで探索的因子分析を実行してみます。1因子から10因子までを指定してやってみましょう。
test3 <- mplusObject(
TITLE = "This is mplusautomation test 3;",
ANALYSIS = "TYPE = EFA 1 10;
ESTIMATOR = MLR;
ROTATION = GEOMIN;
PARALLEL = 50;",
MODEL = "",
VARIABLE = "USEVARIABLES = A1-A5 C1-C5 E1-E5 N1-N5 O1-O5;",
PLOT = "TYPE = PLOT3;",
rdata = bfi)
res3 <- mplusModeler(test3,
dataout = "test3.tsv",
modelout = "test3.inp", run = 1L)結果を見る際には上と同様にsummary関数でも見れますし,以下のように必要な情報だけ引っ張って表にするとみやすくなります。
res3$results$summaries[,c("NumFactors", "ChiSqM_DF", "ChiSqM_Value","ChiSqM_PValue",
"CFI", "TLI", "SRMR", "RMSEA_Estimate", "AIC", "BIC", "aBIC")]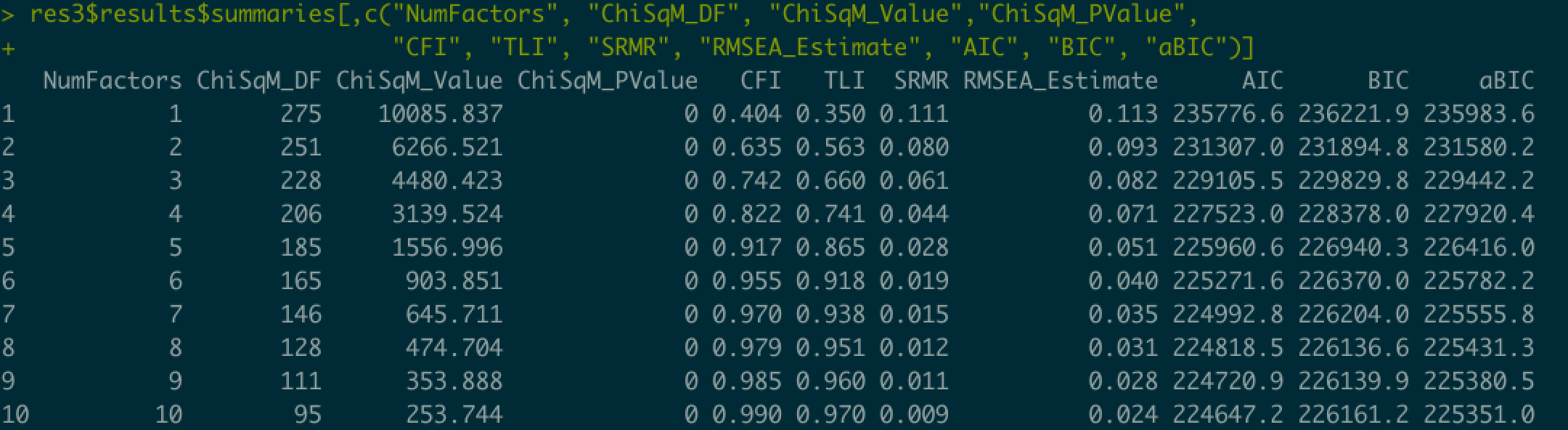
平行分析の結果はgh5というところに入っています。これを取り出せば以下のように平行分析のプロットもできます。
plot(1:25, res3$results$gh5$efa$eigenvalues, type = "b")
points(1:25, res3$results$gh5$efa$parallel_average, type = "b", pch = 16)
points(1:25, res3$results$gh5$efa$parallel_ci, type = "b", pch = 3)
legend("topright", legend = c("sample", "parallel_average", "parallel_95"), pch = c(1,16,3))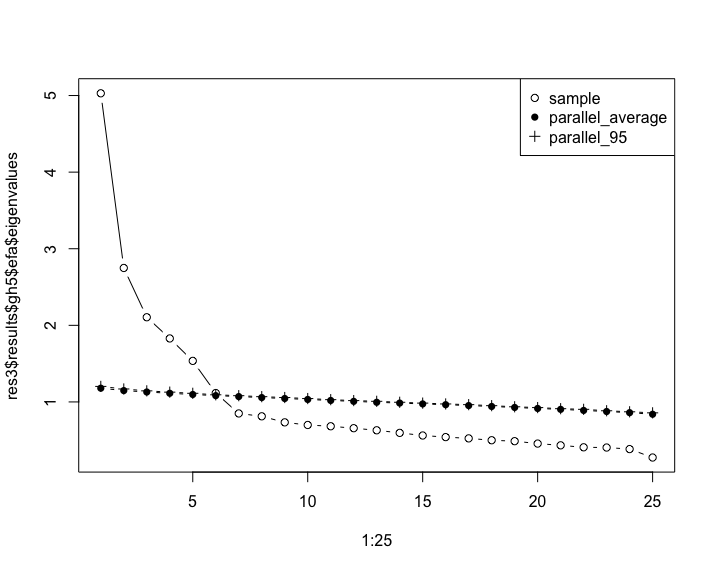
多母集団同時解析の場合
Mplusだと多母集団の同時解析も複雑な制約の掛け方でない限りコマンド1発なので簡単です(もっともRのlavaanでも簡単ですが)。
test4 <- mplusObject(
TITLE = "This is mplusautomation test 4;",
ANALYSIS = "ESTIMATOR = MLR;
MODEL = CONFIGURAL METRIC SCALAR;",
VARIABLE = "USEVARIABLES = A1-A5 C1-C5 E1-E5 N1-N5 O1-O5 GENDER;
GROUPING = gender(1 = MALES, 2 = FEMALES);",
MODEL ="F1 BY A1-A5;
F2 BY C1-C5;
F3 BY E1-E5;
F4 BY N1-N5;
F5 BY O1-O5;",
rdata = bfi)
res4 <- mplusModeler(test4,
dataout = "test4.tsv",
modelout = "test4.inp", run = 1L)
先ほどのように,結果から必要な情報のみ取り出して表にすると便利です。
res4$results$summaries[,c("Model", "ChiSqM_Value", "ChiSqM_DF","ChiSqM_PValue",
"CFI", "SRMR", "RMSEA_Estimate", "AIC", "BIC")]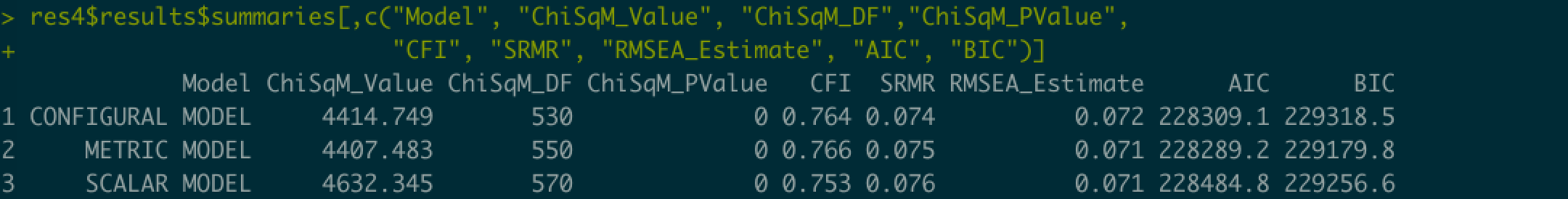
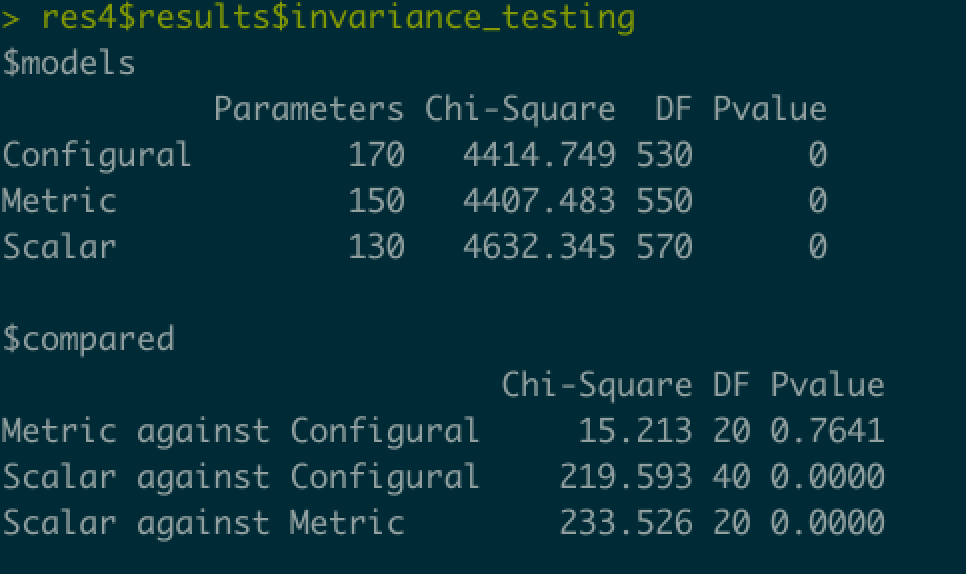
また,結果のオブジェクトのresults$invariance_testingには,カイ二乗値の差の検定の結果が収められています。
Mplus用のデータだけ作りたい
Mplus用のデータだけを作りたい場合にはprepareMplusDataという関数が用意されているようです。
prepareMplusData(bfi, "bfi.tsv")こうするとタブ区切りのファイルが生成されます。欠損地は「.」に置き換えられるようです。なお,inpfile = TRUEと指定すると,元のデータにあったファイル名が入力されたinpファイルも生成してくれますので,それをもとにMplusのsyntaxを書いていくと元のデータと同じ変数名を間違いなく使うことができるようです。keepCols = c("A1", "gender")のようにすると必要な列のみ指定してデータファイルを生成できるみたいです。