lavaanのアウトプットをパス図にする
はじめに
RでSEMをやるときはlavaanをよく使うのですがその結果を綺麗なパス図にしたいということで関数を作りました. lavaanのアウトプットを突っ込むとTikZで描画するためのTeXファイルを吐き出す関数です. 構造方程式を含むもの(フルSEMとか呼ばれるのかな?)には対応しておらず, CFAをそれっぽく出すだけの機能しかありませんが…
インターネットには有難い情報を提供している方がいるもので, 作る際にはbob3さんの以下のブログが参考になりました.
使い方
みんな大好き Holzinger & Swinefordのデータで試してみます. lavaanのアウトプットを関数に突っ込みます. sizeの引数にはTeXの文字サイズの記号を入れると推定値の文字サイズが変えられます.
library(lavaan)
mo <- '
visual =~ x1+x2+x3
textual =~ x4+x5+x6
speed =~ x7+x8+x9
'
res1 <- cfa(mo, HolzingerSwineford1939)
write(lav2tikz(res1, size="scriptsize"), file="res1.tex")作業ディレクトリにres1.texファイルが作られますので, ターミナルでそこまで移動してコンパイルします.
$ latex res1.tex
$ dvipdfmx res1.dvi次のような結果が得られます(webにアップロード用にpngにしていますが).
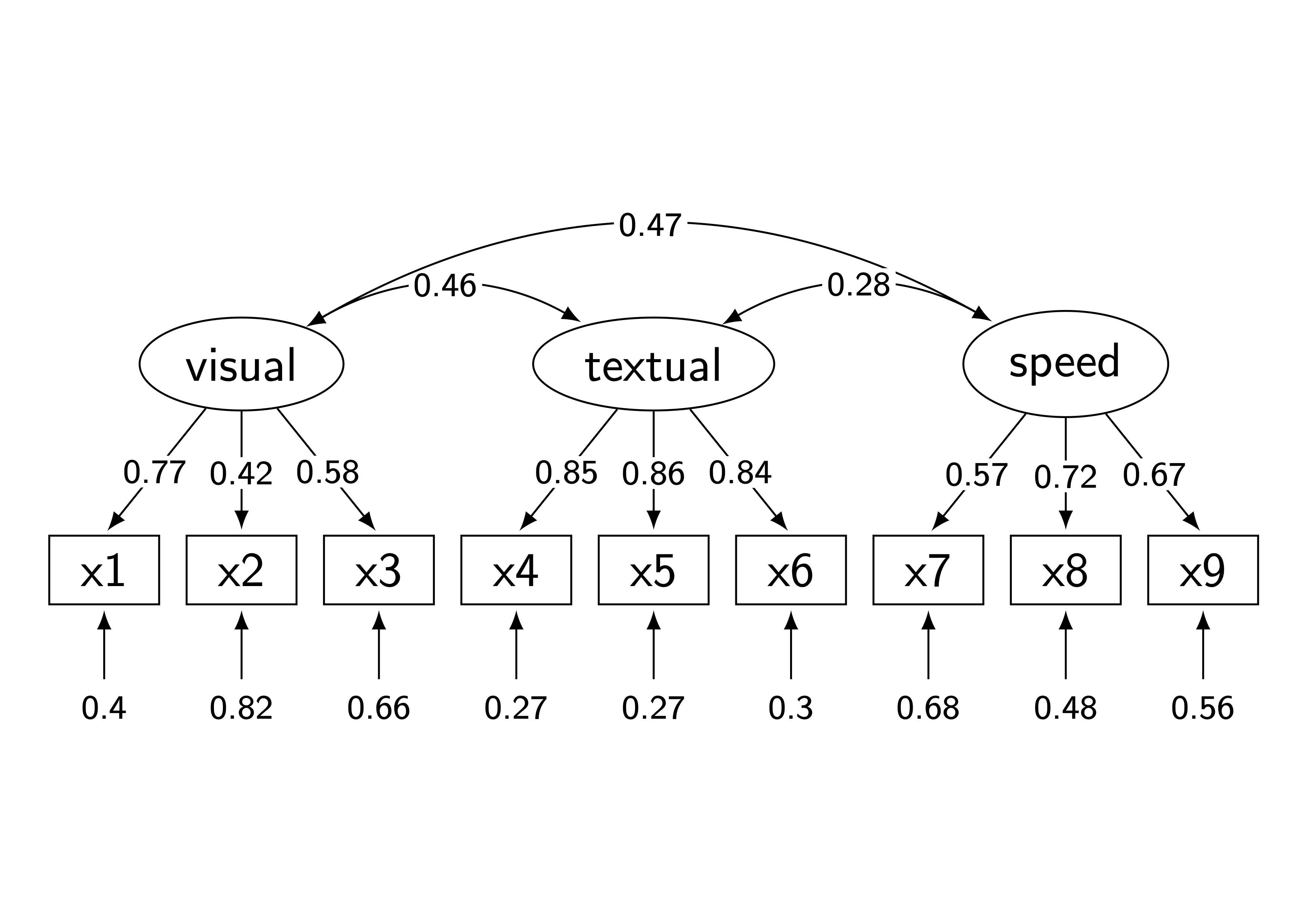
高次因子分析のモデルも一応描画できるようにしています.
mo1 <- '
visual =~ x1+x2+x3
textual =~ x4+x5+x6
speed =~ x7+x8+x9
g =~ visual+textual+speed
'
res2 <- cfa(mo1, data=HolzingerSwineford1939)
write(lav2tikz(res2, size="scriptsize"), file="res2.tex")結果は次の通り.
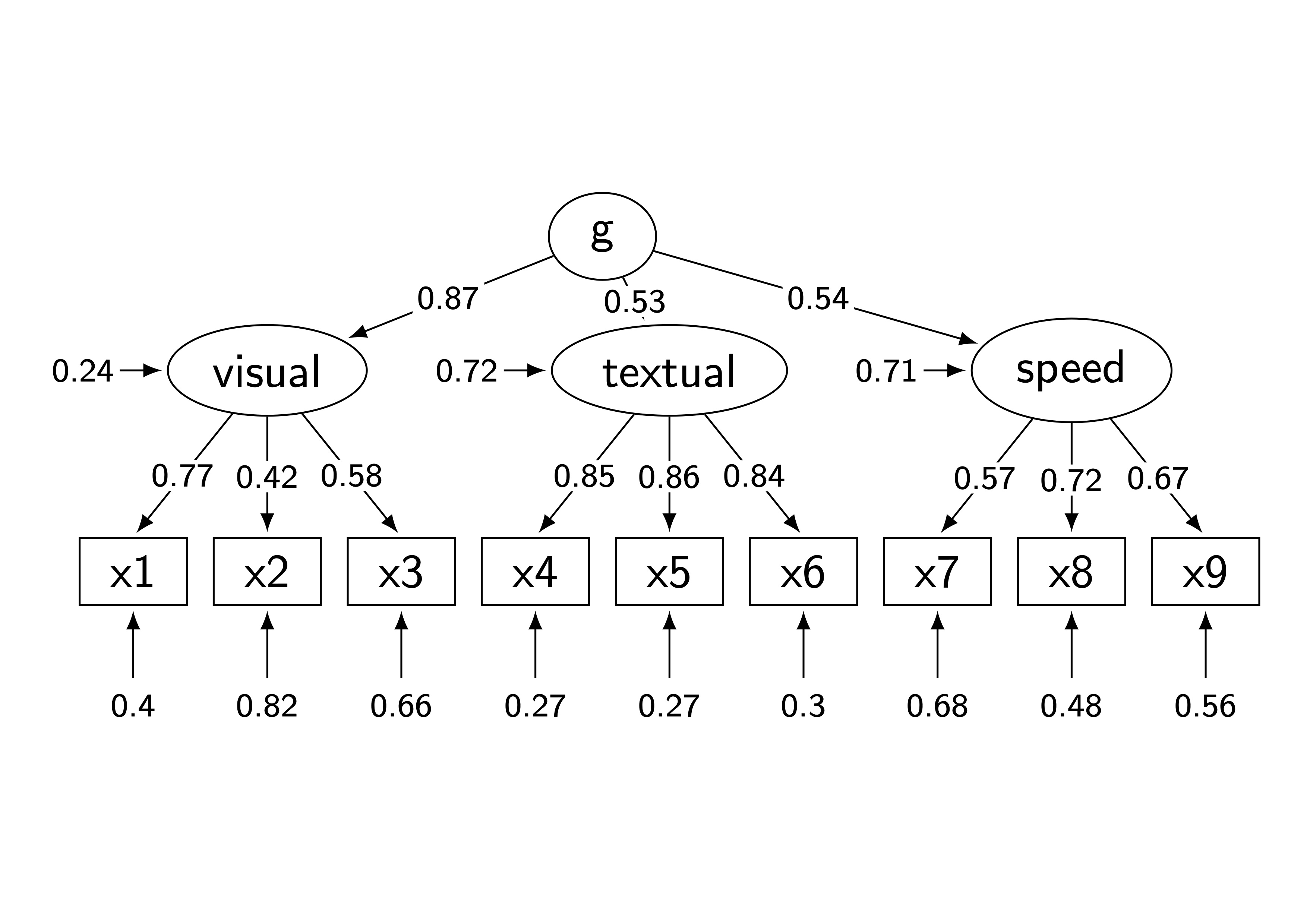
おおむね綺麗に描いてくれますが, gからtextualへのパスの矢印に因子負荷量がかぶっているなど, 一部望んだ結果が得られないところがありますので, 結果をみながらTeXファイルを微調整する必要があります. この場合だとgの位置を高くするか, 推定値の位置を高くするかでしょうか.
ちなみに, 観測変数の並ぶ順番をベクトルで指定できる機能もつけてみました. crossloadingsなどがある場合に思った通りのパス図が得られなかったので.
order <- c("x1","x2", "x3", "x7", "x4", "x5", "x6", "x8", "x9")
write(lav2tikz(res1, size="scriptsize", observed_order=order), file="res3.tex")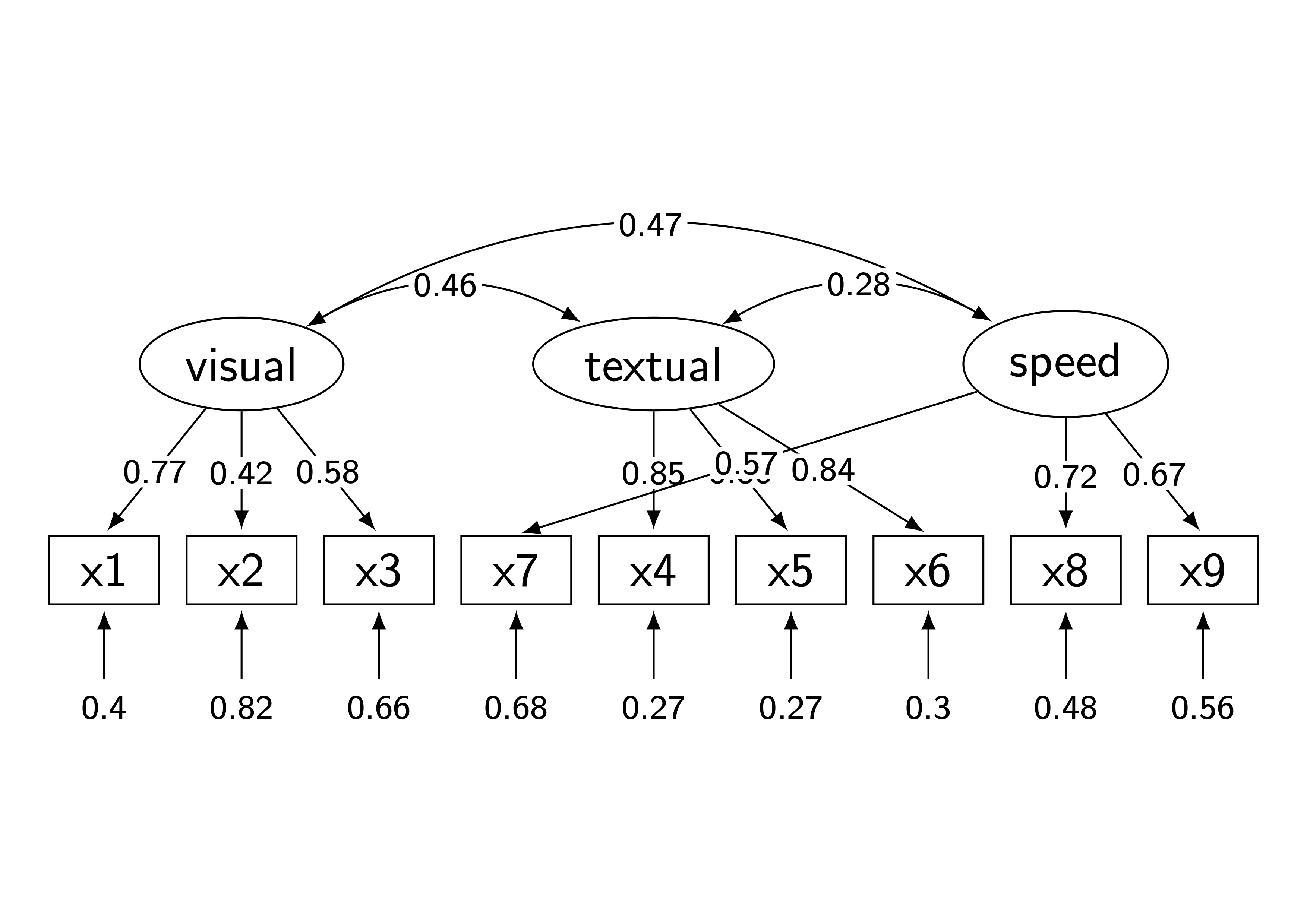
ファイル
以下に置いてあります. 大きいモデルのパス図に対して観測変数を2段に分ける機能をつけようとした残骸が残っていますが, largeの引数をFにしているかぎり悪さはしないはずです… そのうち改修するとは思いますが.
https://github.com/onoshima/myfunction/blob/master/lav2tikz.R
横向きバージョン(2020/02/09追記)
横向きverも作ってみました.
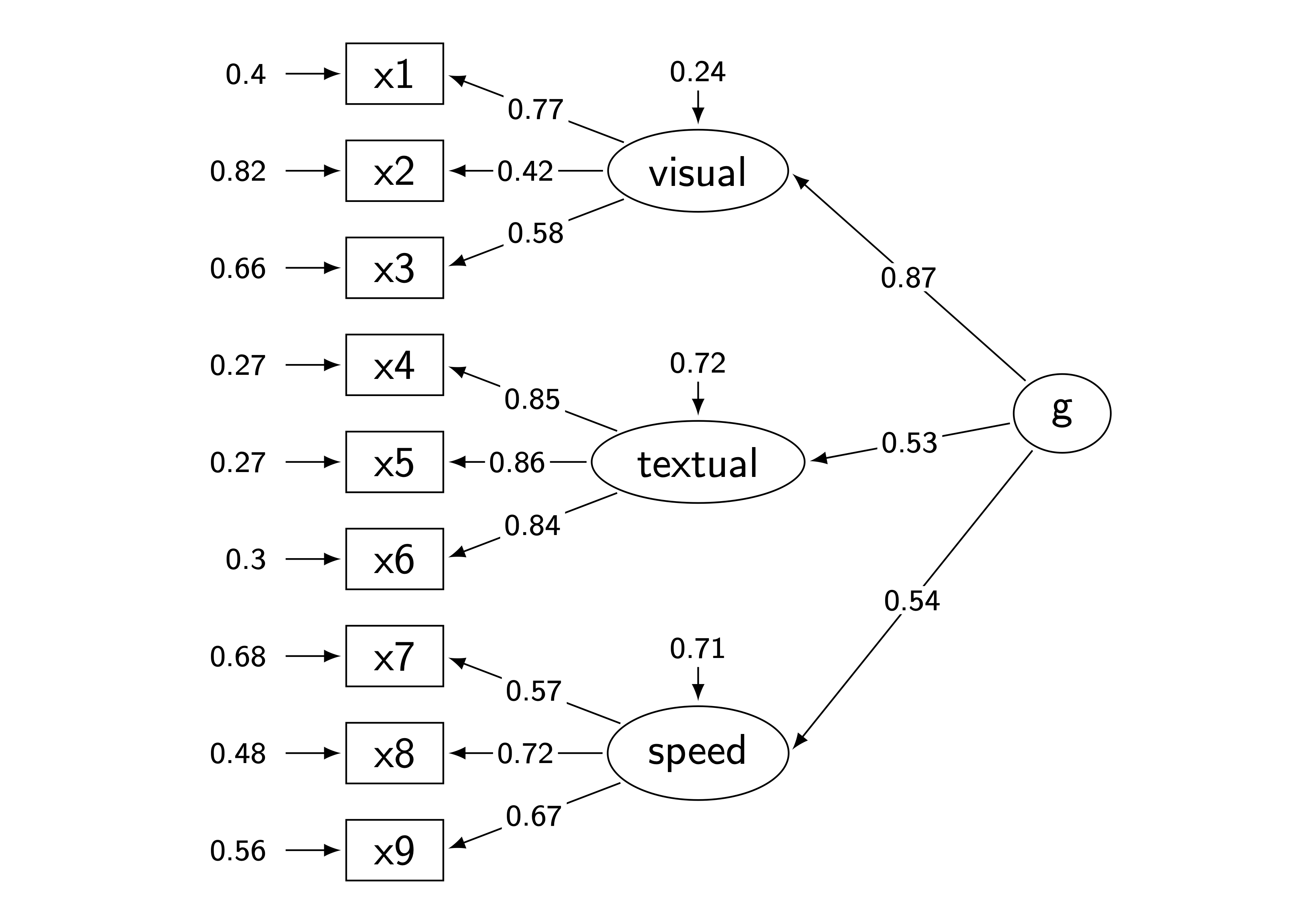
ファイルは以下に置いてあります.
https://github.com/onoshima/myfunction/blob/master/lav2tikz_LR.R