ggstatsplotパッケージについて
Categories:
ggstatsplotパッケージが出たとのことで少しだけ使ってみた記録です。使い方は以下のリンクより。
https://github.com/IndrajeetPatil/ggstatsplot
ggbetweenstats
群間差の比較のためのバイオリンプロットを出すときに使えます。
library(ggplot2)
library(ggstatsplot)
p1 <- ggbetweenstats(data=iris,
x=Species,
y=Sepal.Length,
k=3
)
p1

こんな感じで,検定結果とプロットを一括でやってくれます。便利。 いろいろと細かな設定もできるようです。
p2 <- ggbetweenstats(data=iris,
x=Species,
y=Sepal.Length,
k=2 , #検定結果の小数をどこまで表示するか
title="Distribution of sepal length across Iris species",
plot.type="box", # デフォルトはboxviolin,片方だけにもできる
pairwise.comparisons = F, #多重比較を表示しないこともできる
effsize.type = "biased", #効果量を偏イータにしたければbiase
results.subtitle = F, # FALSEにすると検定結果をサブタイトルから消せる
bf.message = F) # したの方にあるベイズファクターの表示の有無
p2 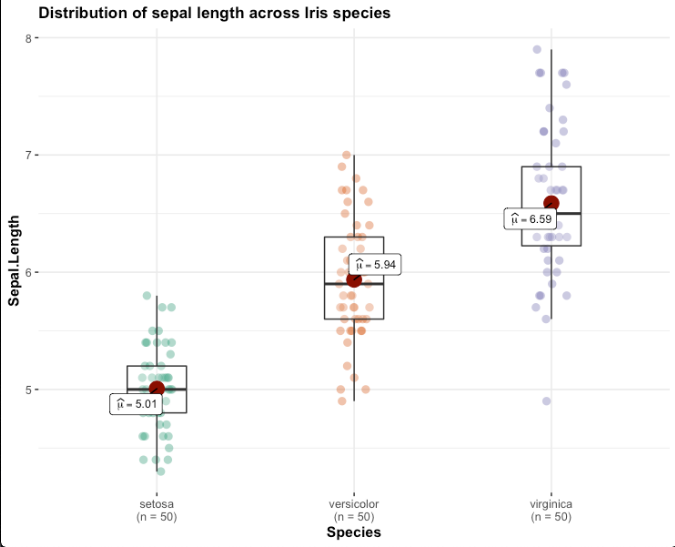
ggscatterstats
散布図+ヒストグラムに無相関の検定を合わせて出力するようです。
p3 <- ggscatterstats(
data = msleep,
x = sleep_rem,
y = awake,
xlab = "REM sleep (in hours)",
ylab = "Amount of time spent awake (in hours)",
title = "Understanding mammalian sleep",
messages = FALSE
)
p3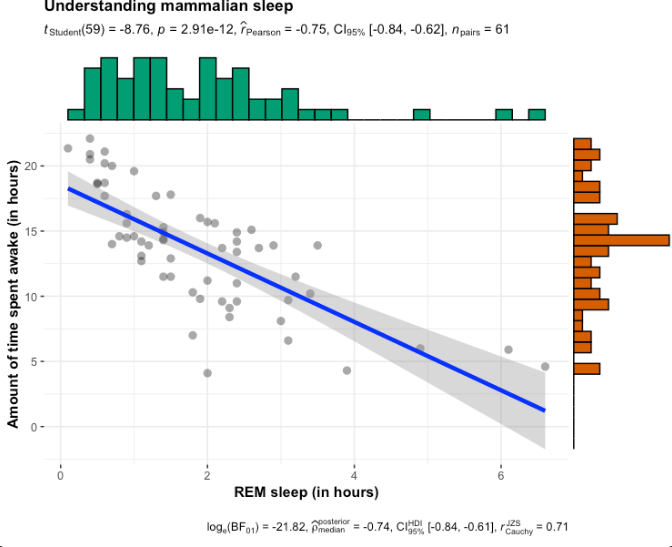
平均(や中央値)の線を引いたり,特定の値を上回る場合にラベルをつけたりできるみたいです。
p4 <- ggscatterstats(
data = msleep,
x = sleep_rem,
y = awake,
xlab = "REM sleep (in hours)",
ylab = "Amount of time spent awake (in hours)",
title = "Understanding mammalian sleep",
messages = FALSE,
centrality.parameter = "mean", #平均値を引く
marginal.type = "density", #ヒストグラムの代わりに密度
label.var= "name", # 各点のラベルとして設定する列名
label.expression = "sleep_rem>6" # ラベルをつける基準
)
p4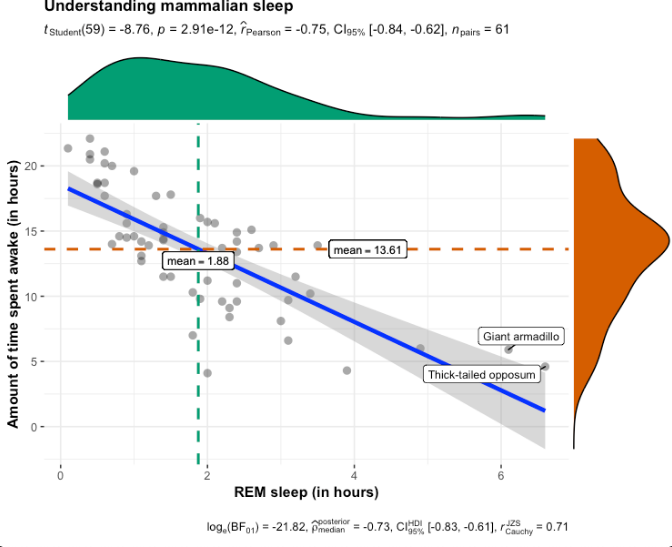
まだまだ他にも機能はたくさんあるのですが,とりあえず試したものだけ。