lapplyおよびparallelパッケージのmclapplyについて調べた際の記録。
lapplyとは
Rで同一の関数を複数のオブジェクトを対象に行うときには, forで繰り返しのループで書くよりも, apply()ファミリーを用いて並列的に処理したほうが早いと言われる。lapplyは, 与えられたリストに対して同一の関数を適用する。lapplyだと戻り値はリストで, sapplyだと戻り値はベクトルである。
例:異なる長さのベクトルの平均
実際に例を見てみると理解が早いかもしれない。長さが10, 100, 1000のベクトルからなるリストそれぞれの平均を求めるには次のように入力する。
x <- list(rnorm(10), rnorm(100), rnorm(1000))
lapply(x, mean) # 戻り値はリスト
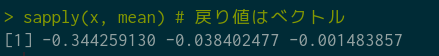
sapply(x, mean) # 戻り値はベクトル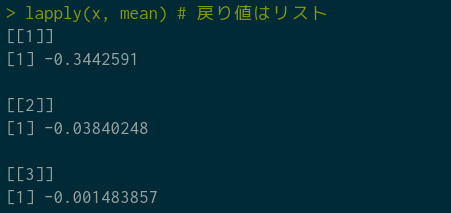
mclapplyとは
lapplyではリストのそれぞれに並列の処理を行うことができることが分かったが, 複数のリストオブジェクトに対して並列的に処理を行いたいという場面もあるかもしれない。そうした際には’parallel’というパッケージの’mclapply’という関数が使えるらしい。
使い方は次のとおり。で繰り返す回数と, 処理する関数を指定するとよいようだ。
replist <- parallel::mclapply(1:1000, function(x){
replicates <- list(mean(rnorm(10)), mean(rnorm(100)),mean(rnorm(1000)))
})戻り値はネストされたリストである。例えば, ネストされたリストを次のように行列の形に直せば, 異なるサンプルサイズにおける標本平均値のばらつきなども分かる。
replicates <- matrix(unlist(replist), 1000, 3, byrow=T)
boxplot(replicates, names=c("size10", "size100", "size1000"))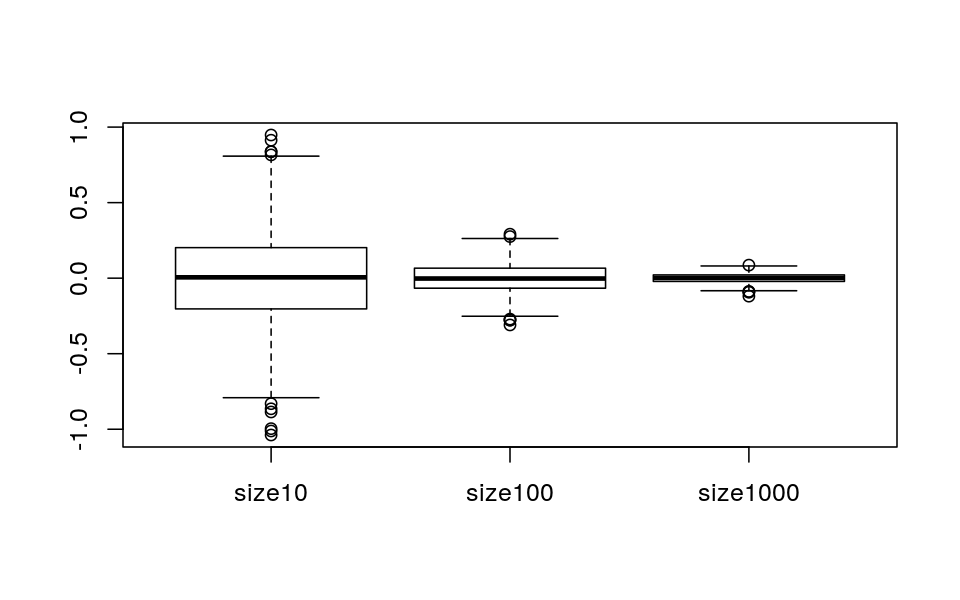
速度の比較
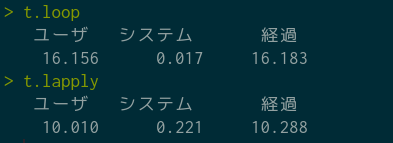
本当にforのループよりも早いか調べてみる。今の処理を100,000回繰り返してみて速度を測ることにする。
t.loop <- proc.time()
x <- list()
for (i in 1:100000){
x[[i]] <- list(mean(rnorm(10)), mean(rnorm(100)), mean(rnorm(1000)))
}
t.loop <- proc.time() - t.loop
t.lapply <- proc.time()
replist <- parallel::mclapply(1:100000, function(x){
replicate <- lapply(list(rnorm(10),rnorm(100), rnorm(1000)),mean)
})
t.lapply <- proc.time() - t.lapply結果は次の通りで, lapplyによる並列処理の方が早いようだ。